李向涛教授指导的2022级博士生于卓含提出了一种跨平台的单细胞聚类方法——基于图嵌入自编码器学习框架,能够准确和有效地聚类细胞和批次效应校正,并揭示了潜在的基因调控机制。该成果以“Topological identification and interpretation for single-cell gene regulation elucidation across multiple platforms using scMGCA”为题发表于国际顶级期刊《Nature Communications》上。
于卓含本科就读于澳门人威尼斯官方数学学院,2020年保研至澳门人威尼斯官方跟随李向涛教授攻读硕士,2022年硕博连读,目前从事单细胞及空间转录组等方面的研究工作。
单细胞RNA测序(scRNA-seq)是一种能够在单个细胞水平上对基因组进行测序分析的技术。相较于传统的测序,单细胞RNA测序能够表征细胞异质性,发现复杂的组织和疾病,并推断单细胞水平的细胞轨迹。其中,对于单细胞数据进行聚类分析是定义细胞类型的关键步骤,也是下游分析的基础。然而,由于RNA的低捕获率和测序深度不足,导致单细胞数据中存在高概率的dropout事件,从而数据呈现高维稀疏的特性,使得聚类分析面临巨大的挑战。
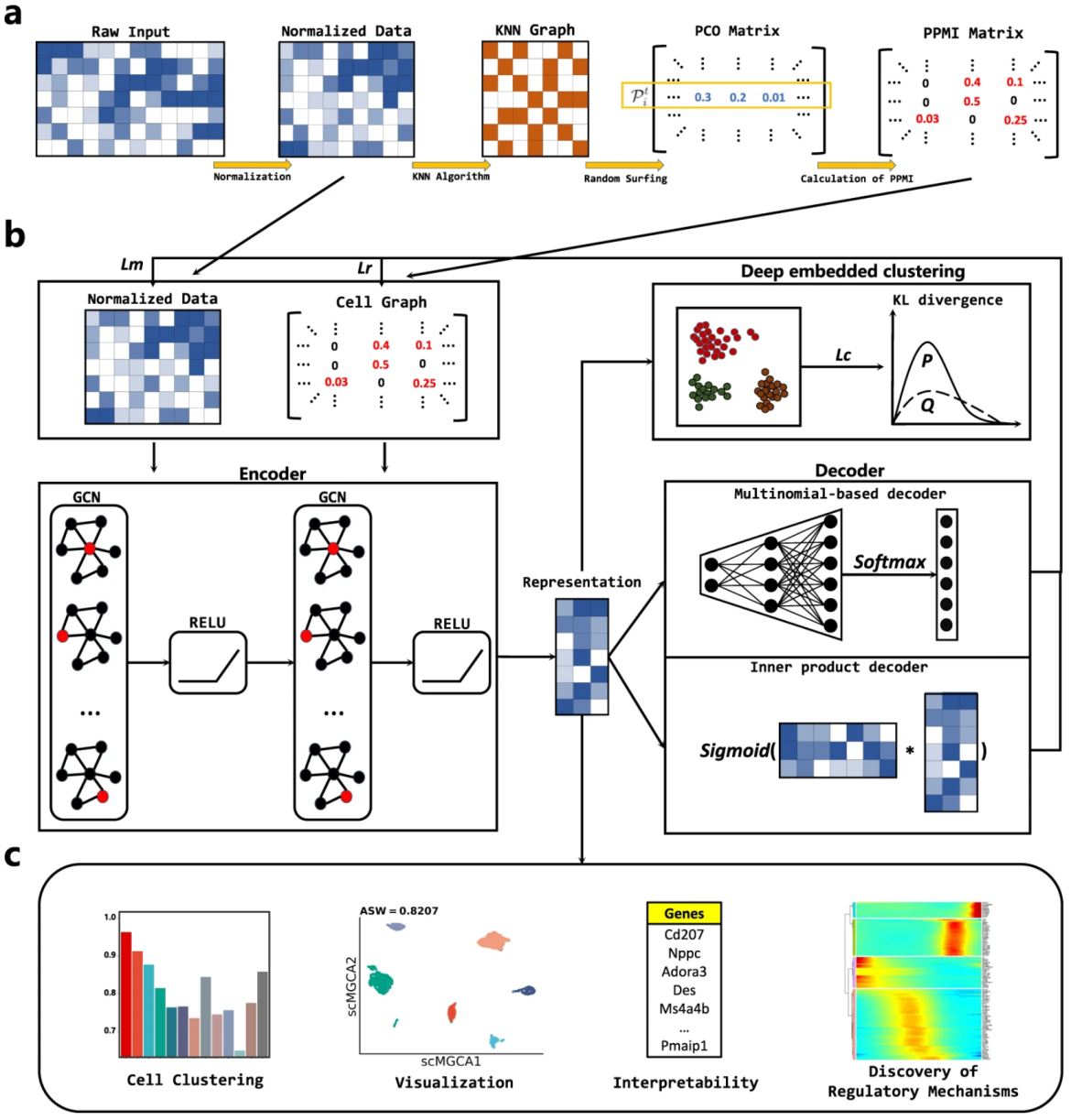
此次李向涛研究组发表于《Nature Communications》的方法基于图自编码器框架学习单细胞数据的图嵌入表示,建模过程分为五个主要的步骤:(1)在归一化的单细胞基因表达矩阵上生成细胞-PPMI矩阵,从而捕捉细胞之间的拓扑结构;(2)通过图卷积网络(GCN)整合基因表达矩阵和细胞-PPMI矩阵,提取主要基因信息和细胞拓扑结构,并将其保存在潜在的嵌入式表示中;(3)采用多项分布模拟单细胞数据的高概率dropout事件,并构建基于多项分布的解码器来表征多模态分布;(4)对嵌入表示进行基于Kullback-Leibler (KL)散度的自优化聚类任务,并与图卷积自编码器中的损失函数同时进行训练;(5)scMGCA学习的潜在嵌入表示可以实现单细胞数据的聚类、降维、可视化和批次效应纠正。
此外,scMGCA还首次在图神经网络框架下对潜在嵌入表示的功能基因组进行了可解释性分析。scMGCA提出了一种基于GCN编码器中参数矩阵标准差的排序筛选方法,从scMGCA的潜在嵌入表示中筛选出可解释的高表达基因。通过对这些基因的功能分析,证明了scMGCA从图神经网络中心隐藏层分析的关键信息具有功能和病理可解释性,为深度图聚类探索生物学可解释性提供了新的途径。
该研究还将 scMGCA 应用于胰腺导管癌(PDAC)数据以进行细胞聚类,并通过细胞簇中的标记基因定义细胞类型。值得注意的是,通过scMGCA 鉴定的 2 型导管细胞都来源于肿瘤细胞。通过进一步的分析,该研究发现 2 型导管细胞富集了与 PDAC 本质上相关的几种路径,其中大多数与临床数据中肿瘤样本所富集的路径一致。这些结果也说明了scMGCA能够阐明复杂疾病的潜在调节机制并提供具有生物学意义的结果。本篇论文是与香港城市大学的Ka-Chun Wong教授共同合作完成。
